Genomomfattande föreningsstudie - Genome-wide association study
Inom genomik är en genomomfattande associeringsstudie ( GWA-studie eller GWAS ), även känd som hela genomassociationsstudie ( WGA-studie eller WGAS ), en observationsstudie av en genomomfattande uppsättning genetiska varianter hos olika individer att se om någon variant är associerad med en egenskap. GWA-studier fokuserar vanligtvis på associationer mellan enkel-nukleotidpolymorfismer (SNP) och egenskaper som stora mänskliga sjukdomar, men kan likaså appliceras på andra genetiska varianter och andra organismer.
När de tillämpas på mänskliga data jämför GWA -studier DNA från deltagare som har varierande fenotyper för en viss egenskap eller sjukdom. Dessa deltagare kan vara personer med en sjukdom (fall) och liknande personer utan sjukdomen (kontroller), eller de kan vara personer med olika fenotyper för ett visst drag, till exempel blodtryck. Detta tillvägagångssätt är känt som fenotyp-först, där deltagarna klassificeras först efter deras kliniska manifestation (er), i motsats till genotyp-först . Varje person ger ett prov av DNA, från vilket miljontals genetiska varianter läses med SNP -matriser . Om en typ av varianten (en allel ) är vanligare hos personer med sjukdomen sägs varianten vara associerad med sjukdomen. De associerade SNP anses sedan markera en region i det mänskliga genomet som kan påverka risken för sjukdom.
GWA-studier undersöker hela genomet, till skillnad från metoder som specifikt testar ett litet antal förspecificerade genetiska regioner. Därför är GWAS en icke-kandidatdriven metod, i motsats till genspecifika kandidatdrivna studier . GWA -studier identifierar SNP och andra varianter i DNA som är associerade med en sjukdom, men de kan inte på egen hand specificera vilka gener som är orsakssamband.
Den första framgångsrika GWAS som publicerades 2002 studerade myokardinfarkt. Denna studiedesign implementerades sedan i landmärket i GWA 2005-studien som undersökte patienter med åldersrelaterad makuladegeneration och fann två SNP: er med signifikant förändrad allelfrekvens jämfört med friska kontroller. Från och med 2017 har över 3000 mänskliga GWA -studier undersökt över 1800 sjukdomar och egenskaper, och tusentals SNP -föreningar har hittats. Förutom när det gäller sällsynta genetiska sjukdomar är dessa föreningar mycket svaga, men även om de kanske inte förklarar mycket av risken, ger de insikt i gener och vägar som kan vara viktiga.
Bakgrund
Alla två mänskliga genomer skiljer sig åt på miljontals olika sätt. Det finns små variationer i de enskilda nukleotiderna i genomerna ( SNP ) såväl som många större variationer, såsom raderingar , infogningar och variationer i kopieringsnummer . Något av dessa kan orsaka förändringar i en individs egenskaper eller fenotyp , som kan vara allt från sjukdomsrisk till fysiska egenskaper som höjd. Runt år 2000, innan GWA -studier introducerades, var den primära metoden för undersökning genom arvsstudier av genetisk koppling i familjer. Detta tillvägagångssätt hade visat sig mycket användbart mot enstaka genstörningar . För vanliga och komplexa sjukdomar visade sig resultaten av genetiska kopplingsstudier dock vara svåra att reproducera. Ett föreslaget alternativ till kopplingsstudier var den genetiska associeringsstudien . Denna studietyp frågar om allelen för en genetisk variant finns oftare än förväntat hos individer med fenotypen av intresse (t.ex. med sjukdomen som studeras). Tidiga beräkningar av statistisk effekt indikerade att detta tillvägagångssätt kan vara bättre än kopplingsstudier för att upptäcka svaga genetiska effekter.
Förutom den konceptuella ramen möjliggjorde flera ytterligare faktorer GWA -studierna. Den ena var tillkomsten av biobanker , som är förvar av mänskligt genetiskt material som kraftigt minskade kostnaden och svårigheten att samla in tillräckligt många biologiska prover för studier. Ett annat var International HapMap Project , som från 2003 identifierade en majoritet av de vanliga SNP: er som förhördes i en GWA -studie. Den haploblock -struktur som identifierades av HapMap -projektet möjliggjorde också fokus på delmängden SNP: er som skulle beskriva det mesta av variationen. Även utvecklingen av metoderna för att genotypa alla dessa SNP: er med hjälp av genotypningssystem var en viktig förutsättning.
Metoder
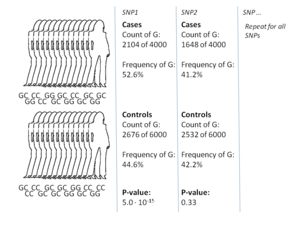
Den vanligaste metoden av GWA studier är fall-kontroll setup, som jämför två stora grupper av individer, en frisk kontrollgruppen och ett fall grupp påverkas av en sjukdom. Alla individer i varje grupp är genotypade för de flesta vanliga kända SNP: erna. Det exakta antalet SNP beror på genotypningstekniken, men är vanligtvis en miljon eller mer. För var och en av dessa SNP undersöks det sedan om allelfrekvensen ändras signifikant mellan fallet och kontrollgruppen. I sådana inställningar är den grundläggande enheten för att rapportera effektstorlekar oddskvoten . Oddskvoten är förhållandet mellan två odds, som i samband med GWA -studier är fallodds för individer som har en specifik allel och oddsen för fall för individer som inte har samma allel.
Anta som ett exempel att det finns två alleler, T och C. Antalet individer i fallgruppen som har allel T representeras av 'A' och antalet individer i kontrollgruppen som har allel T representeras av 'B' . På samma sätt representeras antalet individer i fallgruppen som har allel C med 'X' och antalet individer i kontrollgruppen som har allel C representeras av 'Y'. I detta fall är oddskvoten för allel T A: B (som betyder 'A till B', i standardoddsterminologi) dividerat med X: Y, vilket i matematisk notering är helt enkelt (A/B)/(X/Y).
När allelfrekvensen i fallgruppen är mycket högre än i kontrollgruppen är oddskvoten högre än 1 och vice versa för lägre allelfrekvens. Dessutom beräknas vanligtvis ett P-värde för oddskvotens betydelse med hjälp av ett enkelt chi-kvadrat-test . Att hitta oddskvoter som skiljer sig väsentligt från 1 är syftet med GWA -studien eftersom detta visar att en SNP är associerad med sjukdom. Eftersom så många varianter testas är det vanlig praxis att kräva att p-värdet är lägre än5 × 10 −8 för att betrakta en variant som signifikant.
Det finns flera varianter av detta fallkontrollsätt. Ett vanligt alternativ till fallkontroll-GWA-studier är analys av kvantitativa fenotypiska data, t.ex. höjd eller biomarkörkoncentrationer eller till och med genuttryck . På samma sätt kan alternativ statistik avsedd för dominans eller recessiva penetreringsmönster användas. Beräkningar görs vanligtvis med hjälp av bioinformatikprogram som SNPTEST och PLINK, som också innehåller stöd för många av dessa alternativa statistik. GWAS fokuserar på effekten av enskilda SNP: er. Det är emellertid också möjligt att komplexa interaktioner mellan två eller flera SNP, epistas , kan bidra till komplexa sjukdomar. På grund av det potentiellt exponentiella antalet interaktioner är det både beräkningsmässigt och statistiskt utmanande att upptäcka statistiskt signifikanta interaktioner i GWAS -data. Denna uppgift har hanterats i befintliga publikationer som använder algoritmer inspirerade av data mining. Dessutom försöker forskarna att integrera GWA-data med andra biologiska data, såsom protein-protein-interaktionsnätverk för att extrahera mer informativa resultat.
Ett viktigt steg i majoriteten av GWA -studier är imputering av genotyper vid SNP inte på genotypchipet som används i studien. Denna process ökar kraftigt antalet SNP: er som kan testas för associering, ökar studiens kraft och underlättar metaanalys av GWAS över olika kohorter. Genotypimputation utförs med statistiska metoder som kombinerar GWAS -data tillsammans med en referenspanel med haplotyper. Dessa metoder utnyttjar delning av haplotyper mellan individer under korta sekvenssträckor för att tillskriva alleler. Befintliga mjukvarupaket för genotypimputation inkluderar IMPUTE2, Minimac, Beagle och MaCH.
Förutom beräkningen av associering är det vanligt att ta hänsyn till alla variabler som potentiellt kan förvirra resultaten. Kön och ålder är vanliga exempel på förvirrande variabler. Dessutom är det också känt att många genetiska variationer är associerade med de geografiska och historiska populationer där mutationerna först uppstod. På grund av denna förening måste studier ta hänsyn till deltagarnas geografiska och etniska bakgrund genom att kontrollera vad som kallas befolkningsskiktning . Om de inte gör det kan dessa studier ge falskt positiva resultat.
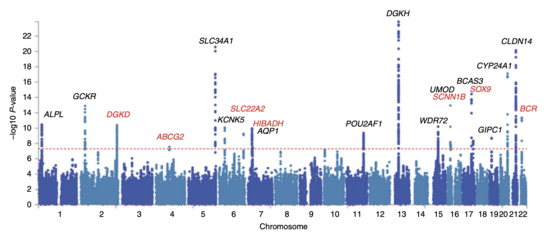
Efter att oddsförhållanden och P-värden har beräknats för alla SNP är ett vanligt tillvägagångssätt att skapa en Manhattan-tomt . I samband med GWA-studier visar denna tomt den negativa logaritmen för P-värdet som en funktion av genomisk plats. Således sticker de SNP: er med den mest betydande kopplingen ut på tomten, vanligtvis som stackar av poäng på grund av haploblock -struktur. Viktigare är att P-värdegränsen för signifikans korrigeras för flera testproblem . Den exakta tröskeln varierar beroende på studie, men den konventionella tröskeln är5 × 10 −8 att vara betydande inför hundratusentals till miljontals testade SNP: er. GWA -studier utför vanligtvis den första analysen i en upptäcktskohort, följt av validering av de mest signifikanta SNP: erna i en oberoende valideringskohort.
Resultat

Försök har gjorts att skapa omfattande kataloger över SNP som har identifierats från GWA -studier. Från och med 2009 är SNP i samband med sjukdomar numrerade i tusentals.
Den första GWA-studien, som genomfördes 2005, jämförde 96 patienter med åldersrelaterad makuladegeneration (ARMD) med 50 friska kontroller. Den identifierade två SNP med signifikant förändrad allelfrekvens mellan de två grupperna. Dessa SNP var belägna i den gen som kodar komplementfaktor H , vilket var ett oväntat fynd i forskningen av ARMD. Resultaten från dessa första GWA -studier har därefter föranlett ytterligare funktionell forskning mot terapeutisk manipulation av komplementsystemet i ARMD. En annan viktig publikation i historien om GWA -studier var Wellcome Trust Case Control Consortium (WTCCC) -studien, den största GWA -studien som någonsin genomförts vid tidpunkten för publiceringen 2007. WTCCC omfattade 14 000 fall av sju vanliga sjukdomar (~ 2 000 individer för var och en av kranskärlssjukdom , typ 1 -diabetes , typ 2 -diabetes , reumatoid artrit , Crohns sjukdom , bipolär sjukdom och högt blodtryck ) och 3000 delade kontroller. Denna studie lyckades med att avslöja många nya sjukdomsgener som ligger bakom dessa sjukdomar.
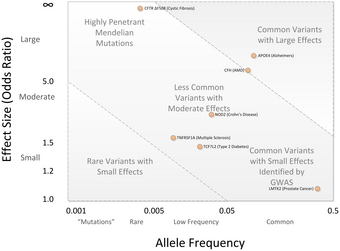
Sedan dessa första landmärken i GWA -studier har det funnits två allmänna trender. Den ena har varit mot större och större provstorlekar. Under 2018 når flera genomomfattande föreningsstudier en total provstorlek på över 1 miljon deltagare, inklusive 1,1 miljoner i en genomomfattande studie av utbildningsnivå och en studie av sömnlöshet som innehåller 1,3 miljoner individer. Anledningen är drivkraften mot att på ett tillförlitligt sätt upptäcka risk-SNP: er som har mindre oddsförhållanden och lägre allelfrekvens. En annan trend har varit mot användning av mer snävt definierade fenotyper, såsom blodlipider , proinsulin eller liknande biomarkörer. Dessa kallas mellanliggande fenotyper , och deras analyser kan vara av värde för funktionell forskning om biomarkörer. En variant av GWAS använder deltagare som är första graders släktingar till personer med en sjukdom. Denna typ av studie har fått namnet genomgående föreningsstudie genom proxy ( GWAX ).
En central diskussionspunkt om GWA -studier har varit att de flesta SNP -variationer som finns i GWA -studier är associerade med endast en liten ökad risk för sjukdomen och har endast ett litet prediktivt värde. Medianoddsförhållandet är 1,33 per risk-SNP, med bara några som visar oddsförhållanden över 3,0. Dessa storheter anses vara små eftersom de inte förklarar mycket av den ärftliga variationen. Denna ärftliga variation uppskattas från ärftlighetsstudier baserade på monozygotiska tvillingar. Till exempel är det känt att 80-90% av höjdvariansen kan förklaras av ärftliga skillnader, men GWA-studier står bara för en minoritet av denna varians.
Kliniska tillämpningar
En utmaning för framtida framgångsrik GWA-studie är att tillämpa resultaten på ett sätt som påskyndar läkemedels- och diagnostikutveckling, inklusive bättre integration av genetiska studier i läkemedelsutvecklingsprocessen och fokus på den genetiska variationens roll för att upprätthålla hälsa som en plan för designa nya läkemedel och diagnostik . Flera studier har undersökt användningen av risk-SNP-markörer som ett sätt att direkt förbättra prognosens noggrannhet . Vissa har funnit att prognosens noggrannhet förbättras, medan andra bara rapporterar mindre fördelar med denna användning. Generellt sett är ett problem med detta direkta tillvägagångssätt den lilla storleken på de observerade effekterna. En liten effekt leder i slutändan till en dålig separation av fall och kontroller och därmed endast en liten förbättring av prognosnoggrannheten. En alternativ tillämpning är därför potentialen för GWA -studier för att belysa patofysiologi .
En sådan framgång är relaterad till att identifiera den genetiska varianten som är associerad med svar på behandling mot hepatit C- virus. För genotyp 1 hepatit C behandlad med Pegylerad interferon-alfa-2a eller Pegylerat interferon-alfa-2b kombinerat med ribavirin , har en GWA-studie visat att SNP nära den humana IL28B- genen, som kodar för interferon lambda 3, är associerade med signifikanta skillnader som svar på behandlingen. En senare rapport visade att samma genetiska varianter också är associerade med den naturliga clearance av genotyp 1 hepatit C -virus. Dessa stora fynd underlättade utvecklingen av personlig medicin och tillät läkare att anpassa medicinska beslut baserat på patientens genotyp.
Målet med att belysa patofysiologi har också lett till ökat intresse för sambandet mellan risk-SNP och genuttryck av närliggande gener, de så kallade expressionskvantitativa egenskaper (eQTL) -studierna. Anledningen är att GWAS-studier identifierar risk-SNP, men inte riskgener, och specifikation av gener är ett steg närmare målbara läkemedelsmål . Som ett resultat inkluderade stora GWA -studier från 2011 vanligtvis omfattande eQTL -analys. En av de starkaste eQTL-effekterna som observerats för ett GWA-identifierat risk-SNP är SORT1-locus. Funktionella uppföljningsstudier av detta lokus med små störande RNA- och gen-knock-out-möss har belyst metabolismen av lågdensitetslipoproteiner , som har viktiga kliniska konsekvenser för hjärt-kärlsjukdom .
Förmaksflimmer
Till exempel avslöjade en metaanalys genomförd 2018 upptäckten av 70 nya loci associerade med förmaksflimmer . Det har identifierats olika varianter associerade med transkriptionsfaktor -kodande-gener, såsom Tbx3 och Tbx5 , NKX2-5 o Pitx2 , vilka är involverade i hjärtöverledning reglering, i jonisk kanalmodulering och hjärtutveckling. Det identifierades också nya gener som är involverade i takykardi ( CASQ2 ) eller associerade med förändring av hjärtmuskelcellkommunikation ( PKP2 ).
Schizofreni
Även om det finns en del forskning som använder en beräkningsmodell med hög precision Protein Interaction Prediction (HiPPIP) som upptäckte 504 nya protein-proteininteraktioner (PPI) associerade med gener kopplade till schizofreni , är bevisen som stöder den genetiska grunden för schizofreni faktiskt kontroversiella och kan lida från några av begränsningen av denna studiemetod.
Jordbruksapplikationer
Växtväxtstadier och avkastningskomponenter
GWA -studier fungerar som ett viktigt verktyg för växtförädling. Med stora genotyp- och fenotypdata är GWAS kraftfulla för att analysera komplexa arvsmetoder för egenskaper som är viktiga avkastningskomponenter som antal korn per spik, vikt av varje spannmål och växtstruktur. I en studie om GWAS i vårvete har GWAS avslöjat en stark korrelation mellan spannmålsproduktion med startdata, biomassa och antal korn per spik.
Växtpatogener
Tillväxten av växtpatogener har inneburit allvarliga hot mot växtens hälsa och biologiska mångfald. Under denna övervägande kan identifiering av vilda typer som har naturligt motstånd mot vissa patogener vara av avgörande betydelse. Dessutom måste vi förutsäga vilka alleler som är associerade med resistensen. GWA-studier är ett kraftfullt verktyg för att upptäcka sambandet mellan vissa varianter och resistensen mot växtpatogenen, vilket är fördelaktigt för att utveckla nya patogenresistenta sorter.
Begränsningar
GWA -studier har flera frågor och begränsningar som kan tas om hand genom korrekt kvalitetskontroll och studieupplägg. Brist på väldefinierade fall- och kontrollgrupper, otillräcklig provstorlek, kontroll för flera tester och kontroll för befolkningsskiktning är vanliga problem. Särskilt den statistiska frågan om flera tester där det har noterats att "GWA-tillvägagångssättet kan vara problematiskt eftersom det massiva antalet utförda statistiska tester har en oöverträffad potential för falskt positiva resultat". Att ignorera dessa korrigerbara problem har citerats som bidragande till en allmän känsla av problem med GWA -metoden. Förutom lätt korrigerbara problem som dessa har några mer subtila men viktiga frågor dykt upp. En högprofilerad GWA-studie som undersökte individer med mycket lång livslängd för att identifiera SNP som är förknippade med livslängd är ett exempel på detta. Publikationen kom att granskas på grund av en skillnad mellan typen av genotypmodell i fallet och kontrollgruppen, vilket gjorde att flera SNP felaktigt framhölls som förknippade med livslängd. Studien drogs sedan tillbaka , men ett modifierat manuskript publicerades senare.
Förutom dessa problem som kan förebyggas har GWA -studier väckt mer grundläggande kritik, främst på grund av deras antagande att gemensam genetisk variation spelar en stor roll för att förklara den ärftliga variationen av vanlig sjukdom. Det har faktiskt uppskattats att för de flesta förhållanden är SNP -ärftligheten som kan hänföras till vanliga SNP: er <0,05. Denna aspekt av GWA -studier har väckt kritik att även om det inte kunde ha varit känt prospektivt, så var GWA -studier i slutändan inte värda utgifterna. GWA -studier möter också kritik mot att den stora variationen av individuella svar eller kompensationsmekanismer till ett sjukdomstillstånd avbryter och maskerar potentiella gener eller orsakssammanhang med sjukdomen. Dessutom identifierar GWA-studier kandidatriskvarianter för befolkningen från vilken deras analys utförs, och med de flesta GWA-studier som härrör från europeiska databaser saknas översättning av de identifierade riskvarianterna till andra icke-europeiska populationer. Alternativa föreslagna strategier innefattar kopplingsanalys . På senare tid, den snabbt sjunkande priset på hela genomet sekvense har också ett realistiskt alternativ till genotypning array baserade GWA studier. Det kan diskuteras om användningen av denna nya teknik fortfarande kallas en GWA-studie, men hög genomströmningssekvensering har potential att vidarebefordra några av bristerna i icke-sekvensering av GWA.
Finkartläggning
Genotypningsarrays utformade för GWAS förlitar sig på kopplingsobalans för att ge täckning av hela genomet genom genotypning av en delmängd av varianter. På grund av detta är det osannolikt att de rapporterade associerade varianterna är de verkliga kausalvarianterna. Associerade regioner kan innehålla hundratals varianter som spänner över stora regioner och omfattar många olika gener, vilket försvårar den biologiska tolkningen av GWAS -loci. Finkartläggning är en process för att förfina dessa listor med associerade varianter till en trovärdig uppsättning som sannolikt innehåller kausalvarianten.
Finkartläggning kräver att alla varianter i den associerade regionen har genotypats eller tillräknats (tät täckning), mycket strikt kvalitetskontroll som resulterar i högkvalitativa genotyper och stora urvalsstorlekar som är tillräckliga för att skilja ut högkorrelerade signaler. Det finns flera olika metoder för att utföra finkartläggning, och alla metoder ger en posterior sannolikhet för att en variant på det locuset är kausal. Eftersom kraven ofta är svåra att tillfredsställa finns det fortfarande begränsade exempel på att dessa metoder tillämpas mer allmänt.
Se även
- Föreningskartläggning
- Epidemiologi
- Gen -miljö interaktion
- Genomik
- Kopplingsobalans
- Molekylär epidemiologi
- Polygen poäng
Referenser
externa länkar
- Genotyp-fenotyp interaktionsprogramvara och databaser på omicX
- Statistiska metoder för analys av genom-breda föreningsstudier [videoföreläsningsserier]
- Hela genomassociationsstudier - av National Human Genome Research Institute
- GWAS Central- en central databas över genetiska associeringsfynd på sammanfattande nivå
- Barrett, Jeff (18 juli 2010). "Hur man läser en genomomfattande föreningsstudie" . Genomen är uppackad.
- Konsortier av genomomfattande associeringsstudier (GWAS)-av Bennett SN, Caporaso, NE, et al.
- PLINK - verktygssats för analys av hela genomförening
- ENCODE threads explorer Effekten av funktionell information för att förstå variation. Nature (tidskrift)