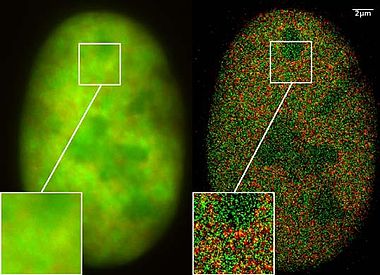
Vertico rumsmodulerad belysning - Vertico spatially modulated illumination
Vertico rumsligt modulerad belysning ( Vertico-SMI ) är det snabbaste ljusmikroskopet för 3D-analys av kompletta celler inom nanometerområdet . Den är baserad på två tekniker som utvecklades 1996, SMI (rumsligt modulerad belysning) och SPDM (spektral precision distansmikroskopi). Den effektiva optiska upplösningen för detta optiska nanoskop har nått närheten till 5 nm i 2D och 40 nm i 3D, vilket kraftigt överstiger λ / 2-upplösningsgränsen (cirka 200 nm för blått ljus) som tillämpas på standardmikroskopi med överföring eller reflektion av naturligt ljus (i motsats till strukturerad belysning eller fluorescens ) enligt Abbe-upplösningsgränsen Den gränsen (även känd som Rayleigh-gränsen ) hade bestämts av Ernst Abbe 1873 och reglerar den uppnåbara upplösningsgränsen för mikroskop med konventionell teknik.
Vertico-SMI-mikroskopet utvecklades av ett team ledt av Christoph Cremer , emeritus vid Heidelberg University , och är baserad på kombinationen av ljusoptiska tekniker för lokaliseringsmikroskopi (SPDM, spektral precision distansmikroskopi ) och strukturerad belysning (SMI, rumsligt modulerad belysning ).
Sedan mars 2008 kan många standard fluorescerande färgämnen som GFP och Alexa fluorescerande färgämnen användas med denna så kallade SPDMphymod (fysiskt modifierbara fluoroforer) lokaliseringsmikroskopi, för vilken endast en enda laservåglängd med lämplig intensitet är tillräcklig för nanoimaging.
Konfiguration
SMI står för en speciell typ av laseroptisk belysning ( rumsligt modulerad belysning ) och Vertico reflekterar det vertikala arrangemanget av mikroskopaxeln som möjliggör analys av fasta celler men också av levande celler med en optisk upplösning under 10 nanometer (1 nanometer = 1 nm = 1 × 10 −9 m).
En särskild egenskap hos denna teknik jämfört med fokuseringstekniker som 4Pi-mikroskopi är exponeringarna för breda fält som gör att hela celler kan avbildas i nanoskalan. En sådan 3D-exponering av en hel cell med en typisk objektstorlek på 20 µm × 20 µm kräver bara 2 minuter. Exponering av breda fält betyder att hela objektet är upplyst och upptäcks samtidigt.
Rumsmodulerad belysning
SMI-mikroskopi är en ljusoptisk process av den så kallade punktspridningsfunktionen - ingenjör. Detta är processer som modifierar punktspridningsfunktionen (PSF) för ett mikroskop på ett lämpligt sätt för att antingen öka den optiska upplösningen, för att maximera precisionen för avståndsmätningar av fluorescerande föremål som är små i förhållande till våglängden för det upplysande ljuset eller extrahera andra strukturella parametrar inom nanometerområdet.
SMI-mikroskopet som utvecklas vid Kirchhoff Institute for Physics vid Heidelberg University uppnår detta på följande sätt: Belysningsintensiteten inom objektintervallet är inte enhetlig, till skillnad från konventionella bredfältsfluorescensmikroskop, men moduleras rumsligt på ett exakt sätt genom användning av två motstående störande laserstrålar längs axeln. Principen för det rumsligt modulerade vågfältet utvecklades 1993 av Bailey et al. SMI-mikroskopi-tillvägagångssättet som används i Heidelberg-applikationen flyttar objektet i steg med hög precision genom vågfältet, eller så flyttas själva vågfältet i förhållande till objektet genom fasförskjutning. Detta resulterar i en förbättrad axiell storlek och avståndsupplösning.
SMI kan kombineras med andra superupplösningstekniker, till exempel med 3D LIMON eller LSI- TIRF som en total intern reflektionsinterferometer med sidostrukturerad belysning. Denna SMI-teknik gjorde det möjligt att förvärva ljusoptiska bilder av autofluoroforfördelningar i sektionerna från mänsklig ögonvävnad med tidigare oöverträffad optisk upplösning. Användning av tre olika exciteringsvåglängder (488, 568 och 647 nm) gör det möjligt att samla in spektralinformation om autofluorescenssignalen. Detta har använts för av mänsklig ögonvävnad som påverkas av AMDs makuladegeneration .
SPDM: lokaliseringsmikroskopi
En enda, liten ljuskälla kan lokaliseras mycket bättre än ett mikroskops upplösning: Även om ljuset kommer att ge en suddig fläck, kan datoralgoritmer användas för att exakt beräkna suddig fläckens centrum , med hänsyn till punktspridningsfunktionen av mikroskopet, detektorns brusegenskaper och så vidare. Detta tillvägagångssätt fungerar emellertid inte när det finns för många källor nära varandra: Källorna suddas ut tillsammans.
SPDM (spektral precision distansmikroskopi) är en familj av tekniker i fluorescensmikroskopi som kringgår detta problem genom att bara mäta några få källor åt gången, så att varje källa är "optiskt isolerad" från de andra (dvs. åtskilda av mer än mikroskopets upplösning, vanligtvis ~ 200-250 nm). Sedan kan ovanstående teknik (hitta mitten av varje suddig plats) användas.
Om molekylerna har en mängd olika spektra (absorptionsspektra och / eller emissionsspektra), är det möjligt att titta på ljus från bara några få molekyler i taget genom att använda lämpliga ljuskällor och filter. Molekyler kan också urskiljas på mer subtila sätt baserat på fluorescerande livslängd och andra tekniker.
Den strukturella upplösningen som kan uppnås med SPDM kan uttryckas i termer av det minsta mätbara avståndet mellan två i deras rumsliga position bestämda punktformiga partiklar med olika spektralegenskaper ("topologisk upplösning"). Modellering har visat att "topologisk upplösning" under lämpliga förhållanden beträffande lokaliseringsprecision, partikeldensitet etc. motsvarar en " rymdfrekvens " som i termer av den klassiska definitionen motsvarar en mycket förbättrad optisk upplösning.
SPDM är en lokaliseringsmikroskopi som uppnår en effektiv optisk upplösning flera gånger bättre än den konventionella optiska upplösningen (ungefär 200-250 nm), representerad av halvbredden av huvudpunkten för den effektiva punktbildfunktionen. Genom att tillämpa lämpliga laseroptiska precisionsprocesser kan position och avstånd betydligt mindre än punktbreddsfunktionens halvbredd (vanligtvis 200-250 nm) mätas med nanometernoggrannhet mellan mål med olika spektralsignaturer. Ett viktigt användningsområde är genomforskning (studie av genomets funktionella organisation ). Ett annat viktigt användningsområde är forskning om membranens struktur.
En av de viktigaste grunderna för lokaliseringsmikroskopi i allmänhet är det första experimentella arbetet för lokalisering av fluorescerande objekt i nanoskalan (3D) 1996 och teoretiskt och experimentellt bevis för lokaliseringsnoggrannhet med synligt ljus i området 1 nm - grunden för lokaliseringsmikroskopi bättre än 1/100 av våglängden.
SPDMphymod: standard fluorescerande färgämnen i blinkande läge som GFP
Först under de senaste två åren har molekyler använts i nanoskopiska studier som avger samma spektrala ljusfrekvens (men med olika spektralsignaturer baserat på blinkande egenskaper) men som kan slås på och av med hjälp av ljus som är nödvändigt för spektral precision avståndsmikroskopi. Genom att kombinera tusentals bilder av samma cell var det möjligt att använda laseroptiska precisionsmätningar för att spela in lokaliseringsbilder med signifikant förbättrad optisk upplösning. Tillämpningen av dessa nya nanoskopiprocesser verkade fram till nyligen mycket svårt eftersom det antogs att endast specialtillverkade molekyler kunde slås på och av på lämpligt sätt genom att använda ljus.
I mars 2008 upptäckte Christoph Cremers laboratorium att detta också var möjligt för många standardfluorescerande färgämnen som GFP , Alexa-färgämnen och fluoresceinmolekyler, förutsatt att vissa fotofysiska förhållanden är närvarande. Med denna så kallade SPDMphymod (fysiskt modifierbara fluoroforer) -teknik är en enda laservåglängd med lämplig intensitet tillräcklig för nano-bild. Däremot behöver andra lokaliseringsmikroskopier två laservåglängder när speciella fotomkopplingsbara / fotoaktiverbara fluorescensmolekyler används.
GFP-genen har introducerats och uttrycks i många prokaryota och eukaryota celler och Nobelpriset i kemi 2008 tilldelades Martin Chalfie , Osamu Shimomura och Roger Y. Tsien för deras upptäckt och utveckling av det gröna fluorescerande proteinet. Fyndet att dessa standard fluorescerande molekyler kan användas utvidgar tillämpningen av SPMD-metoden till många forskningsområden inom biofysik , cellbiologi och medicin .
Standardfluorescerande färgämnen som redan använts framgångsrikt med SPDMphymod-teknologin: GFP, RFP, YFP, Alexa 488, Alexa 568, Alexa 647, Cy2, Cy3, Atto 488 och fluorescein.
LIMON: 3D superupplösningsmikroskopi
LIMON (Light MicrOscopical nanosizing microscopy) uppfanns 2001 vid University of Heidelberg och kombinerar lokaliseringsmikroskopi och rumsligt modulerad belysning till 3D-superupplösningsmikroskopi.
3D-bilderna med Vertico-SMI möjliggörs genom kombinationen av SMI och SPDM, varigenom först SMI och sedan SPDM-processen tillämpas. SMI-processen bestämmer partiklarnas centrum och deras spridning i riktning mot mikroskopaxeln. Medan partiklarnas / molekylernas centrum kan bestämmas med en precision på 1-2 nm, kan spridningen runt denna punkt bestämmas till en axiell diameter på ca. 30-40 nm.
Därefter sidled är positionen för de enskilda partiklar / molekyler bestämdes med användning SPDM, uppnå en precision på några nanometer. För närvarande uppnår SPDM 16 bilder / sek med en effektiv upplösning på 10 nm i 2D (objektplan); ungefär 2000 sådana ramar kombineras med SMI-data (ca 10 sek förvärvstid) för att uppnå en tredimensionell bild med högsta upplösning (effektiv optisk 3D-upplösning ca 40-50 nm). Med en snabbare kamera kan man förvänta sig ännu högre hastigheter (upp till flera hundra bilder / sek, under utveckling). Med lämpliga färgämnen bör ännu högre effektiva optiska 3D-upplösningar vara möjliga
Genom att kombinera SPDMphymod med SMI (båda uppfanns i Christoph Cremers laboratorium 1996) uppnåddes en 3D-dubbelfärgrekonstruktion av de rumsliga arrangemangen för Her2 / neu- och Her3-kluster. Positionerna i proteinklusternas alla tre riktningar kunde bestämmas med en noggrannhet på cirka 25 nm.
Användning av superupplösningsmikroskopi inom industrin
Trots användning i biomedicinska laboratorier kan superupplösningsteknologier fungera som viktiga verktyg inom läkemedelsforskning. De kan vara särskilt användbara vid identifiering och värdering av mål. Till exempel är biomolekylära maskiner (BMM) mycket komplexa nanostrukturer som består av flera stora molekyler och som ansvarar för grundläggande funktioner i kroppscellerna. Beroende på deras funktionella status har de en definierad 3D-struktur. Exempel på biomolekylära maskiner är nukleosomer som gör det möjligt för DNA, en två meter lång bärare av genetisk information, att vika i kroppscellerna i ett utrymme på några miljonedel av en millimeter i diameter. Därför kan DNA fungera som ett informations- och kontrollcenter.
Genom att använda LIMON 3D i kombination med LIMON-komplexmärkning är det för första gången möjligt att synliga proteiner eller nukleinsyror från ett 3D-molekylkomplex av de så kallade biomolekylära maskinerna syns utan att förstöra komplexet. Hittills var problemet i de flesta fall att komplexet måste förstöras för detaljerad analys av de enskilda makromolekylerna däri. Alternativt användes virtuella datorsimuleringsmodeller eller dyra kärnmagnetiska resonansmetoder för att visualisera den tredimensionella strukturen hos sådana komplex.
Litteratur
externa länkar
- GFP Superresolution
- Publikationslista optisk nanoskopi
- pressmeddelande från universitetet i Heidelberg