Virusklassificering - Virus classification
Virusklassificering är processen att namnge virus och placera dem i ett taxonomiskt system som liknar klassificeringssystemen som används för cellulära organismer .
Virus klassificeras efter fenotypiska egenskaper, såsom morfologi , typ av nukleinsyra , replikationssätt, värdorganismer och vilken typ av sjukdom de orsakar. Den formella taxonomiska klassificeringen av virus är ansvarig för International Committee on Taxonomy of Virus (ICTV), även om Baltimores klassificeringssystem kan användas för att placera virus i en av sju grupper baserat på deras sätt att mRNA -syntes. Specifika namnkonventioner och ytterligare klassificeringsriktlinjer anges av ICTV.
En katalog med alla världens kända virus har föreslagits och 2013 påbörjades några preliminära insatser.
Virusart definition
Arter utgör grunden för alla biologiska klassificeringssystem. Före 1982 trodde man att virus inte kunde fås att passa Ernst Mayrs reproduktiva koncept av arter, och därför inte var mottagliga för sådan behandling. 1982 började ICTV definiera en art som "ett kluster av stammar" med unika identifierande egenskaper. År 1991 antogs den mer specifika principen att en virusart är en polyetisk klass av virus som utgör en replikerande släktlinje och upptar en viss ekologisk nisch.
I juli 2013 ändrades ICTV -definitionen av arter till följande: "En art är en monofyletisk grupp av virus vars egenskaper kan särskiljas från andra arters egenskaper genom flera kriterier." Dessa kriterier inkluderar kapsidens struktur , förekomsten av ett hölje, genuttrycksprogrammet för dess proteiner, värdintervall, patogenicitet och viktigast av allt genetisk sekvenslikhet och fylogenetiskt förhållande.
De faktiska kriterierna som används varierar beroende på taxon och kan ibland vara inkonsekventa (godtyckliga tröskelvärden för likhet) eller ibland inte relaterade till härstamning (geografi). Frågan är för många ännu inte klar.
ICTV -klassificering
Den internationella kommittén för taxonomi för virus började utarbeta och införa regler för namngivning och klassificering av virus tidigt på 1970 -talet, en ansträngning som fortsätter fram till idag. ICTV är det enda organ som belastas av International Union of Microbiological Societies med uppgiften att utveckla, förfina och upprätthålla en universell virustaxonomi. Systemet delar många funktioner med klassificeringssystemet för cellulära organismer , till exempel taxonstruktur . Vissa skillnader finns dock, till exempel universell användning av kursiv för alla taxonomiska namn, till skillnad från i den internationella nomenklaturkoden för alger, svampar och växter och International Code of Zoological Nomenclature .
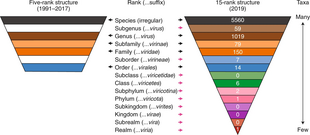
Viral klassificering börjar på rikets nivå och fortsätter enligt följande, med de taxonomiska suffixen inom parentes:
- Realm ( -viria )
Till skillnad från systemet med binomial nomenklatur som används i cellarter finns det för närvarande ingen standardiserad form för virusartnamn. För närvarande kräver ICTV att ett artnamn måste innehålla så få ord som möjligt medan det förblir distinkt och inte bara måste innehålla ordet virus och värdnamnet. Artnamn har ofta formen av [Disease] -virus , särskilt för högre växter och djur. År 2019 publicerade ICTV ett förslag om att anta ett mer formaliserat system för binomial nomenklatur för virusartnamn som ska röstas om 2020. Vissa virologer protesterade senare mot den potentiella namnsystemändringen och hävdade att debatten kom medan många i fältet var upptaget på grund av COVID-19-pandemin .
Från och med 2019 används alla nivåer av taxa utom subrealm, subkingdom och subclass. Fyra världar, en incertae sedis -ordning, 24 incertae sedis -familjer och tre incertae sedis -släkten känns igen:
Riken : Duplodnaviria , Monodnaviria , Adnaviria , Ribozyviria , Riboviria och Varidnaviria
Incertae sedis -familjer :
- Alphasatellitidae
- Ampullaviridae
- Anelloviridae
- Avsunviroidae
- Baculoviridae
- Bicaudaviridae
- Clavaviridae
- Finnlakeviridae
- Fuselloviridae
- Globuloviridae
- Guttaviridae
- Halspiviridae
- Hytrosaviridae
- Nimaviridae
- Nudiviridae
- Ovaliviridae
- Plasmaviridae
- Polydnaviridae
- Portogloboviridae
- Pospiviroidae
- Spiraviridae
- Thaspiviridae
- Tolecusatellitidae
- Tristromaviridae
Incertae sedis genera : Deltavirus , Dinodnavirus , Rhizidiovirus
Strukturbaserad virusklassificering
Det har föreslagits att likhet i virionsmontering och struktur som observerats för vissa virala grupper som infekterar värdar från olika livsområden (t.ex. bakteriella tektivirus och eukaryota adenovirus eller prokaryota Caudovirales och eukaryota herpesvirus) återspeglar ett evolutionärt förhållande mellan dessa virus. Därför har strukturella förhållanden mellan virus föreslagits användas som grund för att definiera taxa på högre nivå-strukturbaserade virala linjer-som kan komplettera ICTV-klassificeringsschemat för 2010.
ICTV har successivt lagt till många taxa på högre nivå med hjälp av relationer i proteinveck. Alla fyra områden som definieras i 2019 års utgåva definieras av närvaron av ett protein från en viss strukturell familj.
Baltimore klassificering

Baltimore-klassificering (definierades först 1971) är ett klassificeringssystem som placerar virus i en av sju grupper beroende på en kombination av deras nukleinsyra ( DNA eller RNA ), stränghet (enkelsträngad eller dubbelsträngad), känsla och metod för replikering . Uppkallad efter David Baltimore , en Nobelprisvinnande biolog, betecknas dessa grupper med romerska siffror . Andra klassificeringar bestäms av sjukdomen som orsakas av viruset eller dess morfologi, och ingen av dem är tillfredsställande på grund av att olika virus antingen orsakar samma sjukdom eller ser väldigt lika ut. Dessutom är virusstrukturer ofta svåra att bestämma under mikroskopet. Att klassificera virus efter deras genom innebär att de i en viss kategori alla kommer att bete sig på ett liknande sätt, vilket ger en indikation på hur man ska gå vidare med vidare forskning. Virus kan placeras i en av de sju följande grupperna:
- I: dsDNA -virus (t.ex. adenovirus , herpesvirus , poxvirus )
- II: ssDNA -virus (+ sträng eller "känsla") DNA (t.ex. Parvovirus )
- III: dsRNA -virus (t.ex. reovirus )
- IV: (+) ssRNA -virus (+ sträng eller känsla) RNA (t.ex. Coronaviruses , Picornaviruses , Togaviruses )
- V: ( -) ssRNA -virus ( - sträng eller antisense) RNA (t.ex. Orthomyxovirus , Rhabdovirus )
- VI: ssRNA-RT-virus (+ sträng eller känsla) RNA med DNA-intermediär i livscykeln (t.ex. retrovirus )
- VII: dsDNA-RT-virus DNA med RNA-intermediär i livscykeln (t.ex. hepadnavirus )
DNA -virus
Virus med ett DNA -genom , med undantag för DNA -omvänd transkriberande virus , är medlemmar i tre av de fyra erkända virala sfärerna : Duplodnaviria , Monodnaviria och Varidnaviria . Men incertae sedis order Ligamenvirales , och många andra incertae sedis familjer och släkten, används också för att klassificera DNA-virus. Domänerna Duplodnaviria och Varidnaviria består av dubbelsträngade DNA-virus; andra dubbelsträngade DNA-virus är incertae sedis . Domänen Monodnaviria består av enkelsträngade DNA-virus som generellt kodar för ett HUH- endonukleas ; andra enkelsträngade DNA-virus är incertae sedis .
- Grupp I : virus har dubbelsträngat DNA. Virus som orsakar vattkoppor och herpes finns här.
- Grupp II : virus har enkelsträngat DNA.
| Virusfamilj | Exempel (vanliga namn) | Virion naken/innesluten |
Capsid symmetri |
Nukleinsyratyp | Grupp |
|---|---|---|---|---|---|
| 1. Adenoviridae | Hundhepatitvirus , vissa typer av förkylning | Naken | Icosahedral | ds | I |
| 2. Papovaviridae | JC -virus , HPV | Naken | Icosahedral | ds cirkulär | I |
| 3. Parvoviridae | Humant parvovirus B19 , parvovirus hos hundar | Naken | Icosahedral | ss | II |
| 4. Herpesviridae | Herpes simplex-virus , varicella-zoster-virus , cytomegalovirus , Epstein-Barr-virus | Omslagen | Icosahedral | ds | I |
| 5. Poxviridae | Smittkoppor , koxor , myxomvirus , aporoxor , vacciniavirus | Komplexa rockar | Komplex | ds | I |
| 6. Anelloviridae | Torque teno -virus | Naken | Icosahedral | ss cirkulär | II |
| 7. Pleolipoviridae | HHPV1 , HRPV1 | Omslagen | ss/ds linjär/cirkulär | I/II |
RNA -virus
Alla virus som har ett RNA- genom , och som kodar för ett RNA-beroende RNA-polymeras (RdRp), är medlemmar i riket Orthornavirae , inom riket Riboviria .
- Grupp III : virus har dubbelsträngade RNA-genomer, t.ex. rotavirus .
- Grupp IV : virus har positiva sinnes enkelsträngade RNA-genomer. Många kända virus finns i denna grupp, inklusive picornavirus (som är en familj av virus som inkluderar välkända virus som hepatit A-virus, enterovirus, rinovirus, poliovirus och mul- och klövsjuka), SARS- virus, hepatit C -virus, gula feberviruset och röda hundvirus.
- Grupp V : virus besitter negativkänsliga enkelsträngade RNA-genomer. Ebola- och Marburg -virus är välkända medlemmar i denna grupp, tillsammans med influensavirus , mässling , påssjuka och rabies .
Omvänd transkriberande virus
Alla virus som kodar för ett omvänt transkriptas (även känt som RT- eller RNA-beroende DNA-polymeras) är medlemmar i klassen Revtraviricetes , inom phylum Arterviricota , Kingdom Pararnavirae och Riboviria . Klassen Blubervirales innehåller den enda familjen Hepadnaviridae av DNA RT (omvänt transkriberande) virus; alla andra RT -virus är medlemmar i klassen Ortervirales .
- Grupp VI : virus har ensträngade RNA-virus som replikeras genom en DNA-mellanprodukt. De retrovirus ingår i denna grupp, där HIV är en medlem.
- Grupp VII : virus har dubbelsträngade DNA-genomer och replikerar med omvänt transkriptas. Den hepatit B- virus kan hittas i denna grupp.
| Virusfamilj | Exempel (vanliga namn) | Capsid naken/kuvert |
Capsid symmetri |
Nukleinsyratyp | Grupp |
|---|---|---|---|---|---|
| 1. Retroviridae | HIV | Omslagen | dimer -RNA | VI | |
| 2. Caulimoviridae | Caulimovirus , Cacao swollen-shoot virus (CSSV) | Naken | VII | ||
| 3. Hepadnaviridae | Hepatit B -virus | Omslagen | Icosahedral | cirkulär, delvis ds | VII |
Historiska system
Holmes klassificering
Holmes (1948) använde en Linnéaxonomi med binomial nomenklatur för att klassificera virus i 3 grupper under en ordning, Virales . De placeras enligt följande:
- Grupp I: Phaginae (angriper bakterier)
- Grupp II: Phytophaginae (attackerar växter)
- Grupp III: Zoophaginae (attackerar djur)
Systemet accepterades inte av andra på grund av dess försummelse av morfologiska likheter.
Subvirala medel
Följande smittämnen är mindre än virus och har bara några av deras egenskaper. Sedan 2015 har ICTV tillåtit dem att klassificeras på ett liknande sätt som virus är.
Viroider och virusberoende medel
Viroider
- Familj Avsunviroidae
- Släktet Avsunviroid ; typart : Avocado sunblotch viroid
- Släktet Pelamoviroid ; typart: Persika latent mosaikviroid
- Släktet Elaviroid ; typart: Aubergine latent viroid
- Familj Pospiviroidae
- Släktet Pospiviroid ; typart: Potatisspindelknöl viroid
- Släktet Hostuviroid ; typart: Hoppstunt viroid
- Släktet Cocadviroid ; typart: Coconut cadang-cadang viroid
- Släktet Apscaviroid ; typart: Äpple ärr hud viroid
- Släktet Coleviroid ; typart: Coleus blumei viroid 1
Satelliter
Satelliter är beroende av saminfektion av en värdcell med ett hjälpvirus för produktiv multiplikation. Deras nukleinsyror har väsentligen distinkta nukleotidsekvenser från antingen deras hjälparvirus eller värd. När ett satellit -subviralt medel kodar kappproteinet i vilket det är inkapslat, kallas det sedan ett satellitvirus.
Satellitliknande nukleinsyror liknar satellitnukleinsyror genom att de replikeras med hjälp av hjälpvirus. Men de skiljer sig åt genom att de kan koda funktioner som kan bidra till framgången för deras hjälpvirus; medan de ibland anses vara genomiska element i deras hjälparvirus, finns de inte alltid inom deras hjälparvirus.
- Satellitvirus
- Ensträngade RNA- satellitvirus
- (namnlös familj)
- Familj Sarthroviridae
- Macronovirus - Macrobrachium satellitvirus 1 (extra litet virus)
- (namnlöst släkte) - Nilaparvata lugens commensal X -virus
- (namnlöst släkte) -Kroniskt bi-förlamning-satellitvirus
- Dubbelsträngade DNA- satellitvirus
- Familj Lavidaviridae - Virophages
- Ensträngade DNA- satellitvirus
- Släkte Dependoparvovirus - adenoassocierat virus grupp
- Ensträngade RNA- satellitvirus
- Satellitnukleinsyror
- Ensträngade satellit-DNA
- Familj Alphasatellitidae (kodar för ett replikationsinitieringsprotein)
- Familj Tolecusatellitidae (kodar en patogenicitetsdeterminant βC1)
- Dubbelsträngade satellit-RNA
- Ensträngade satellit-RNA
- Undergrupp 1: Stora satellit -RNA
- Undergrupp 2: Små linjära satellit -RNA
- Undergrupp 3: Cirkulära satellit -RNA ( virusoider )
- Släktet Deltavirus
- Polerovirus -associerade RNA
- Satellitliknande RNA
- Satellitliknande DNA
- Ensträngade satellit-DNA
Defekta störande partiklar
Defekta störande partiklar är defekta virus som har förlorat sin förmåga att replikera utom i närvaro av ett hjälpvirus, som normalt är föräldraviruset. De kan också störa hjälparviruset.
- Defekta störande partiklar (RNA)
- Defekta störande partiklar (DNA)
Se även
Anteckningar
- ^ Zimmer C (5 september 2013). "En katalog för alla världens virus?" . New York Times . Hämtad 6 september 2013 .
- ^ a b Alimpiev, Egor (15 mars 2019). Ompröva konceptet virusarter (PDF) .
- ^ Adams MJ, Lefkowitz EJ, King AM, Carstens EB (december 2013). "Nyligen överenskomna ändringar av den internationella koden för virusklassificering och nomenklatur" . Arkiv för virologi . 158 (12): 2633–9. doi : 10.1007/s00705-013-1749-9 . PMID 23836393 .
- ^ "International Committee on Taxonomy of Viruses (ICTV)" . Internationella kommittén för taxonomi för virus (ICTV) . Hämtad 2021-06-10 .
- ^ Peterson, A Townsend (23 juli 2014). "Definiera virala arter: göra taxonomi användbar" . Virology Journal . 11 (1): 131. doi : 10.1186/1743-422X-11-131 . PMC 4222810 . PMID 25055940 .
- ^ Lefkowitz EJ, Dempsey DM, Hendrickson RC, Orton RJ, Siddell SG, Smith DB (januari 2018). "Virustaxonomi: databasen från International Committee on Taxonomy of Virus (ICTV)" . Nukleinsyror . 46 (D1): D708 – D717. doi : 10.1093/nar/gkx932 . PMC 5753373 . PMID 29040670 .
- ^ a b "ICTV -kod" . talk.ictvonline.org . Internationella kommittén för taxonomi för virus . Hämtad 26 april 2020 .
- ^ "The International Code of Virus Classification and Nomenclature" . ICTV . Internationella kommittén för taxonomi för virus . Hämtad 2 september 2020 .
- ^ Siddell, Stuart; Walker, Peter; Lefkowitz, Elliot; Mushegian, Arcady; Dutilh, Bas; Harrach, Balázs; Harrison, Robert; Junglen, Sandra; Knowles, Nick; Kropinski, Andrew; Krupovic, Mart; Kuhn, Jens; Nibert, Max; Rubino, Luisa; Sabanadzovic, Sead; Simmonds, Peter; Varsani, Arvind; Zerbini, Francisco; Davison, Andrew (3 december 2019). "Binomial nomenklatur för virusarter: ett samråd" . Arch Virol . 165 (2): 519–525. doi : 10.1007/s00705-019-04477-6 . PMC 7026202 . PMID 31797129 .
- ^ Mallapaty, Smriti (30 juli 2020). "Bör regler för namngivning av virus ändras under en pandemi? Frågan delar virologer" . Natur . 584 (7819): 19–20. Bibcode : 2020Natur.584 ... 19M . doi : 10.1038/d41586-020-02243-2 . PMID 32733098 .
- ^ a b "Virustaxonomi: 2019 -utgåva" . talk.ictvonline.org . Internationella kommittén för taxonomi för virus . Hämtad 26 april 2020 .
- ^ Bamford DH (maj 2003). "Bildar virus släktlinjer på olika livsområden?". Forskning i mikrobiologi . 154 (4): 231–6. doi : 10.1016/S0923-2508 (03) 00065-2 . PMID 12798226 .
- ^ Krupovič M, Bamford DH (december 2010). "Ordning till det virala universum" . Journal of Virology . 84 (24): 12476–9. doi : 10.1128/JVI.01489-10 . PMC 3004316 . PMID 20926569 .
- ^ "Virustaxonomi: 2019 -utgåva" . talk.ictvonline.org . Internationella kommittén för taxonomi för virus . Hämtad 26 april 2020 .
- ^ "Baltimore klassificering av virus" (webbplats.) Molekylärbiologi webbbok - http://web-books.com/ . Hämtad 2008-08-18.
- ^ "Virustaxonomi: 2019 -utgåva" . talk.ictvonline.org . Internationella kommittén för taxonomi för virus . Hämtad 25 april 2020 .
- ^ Koonin EV, Dolja VV, Krupovic M, Varsani A, Wolf YI, Yutin N, Zerbini M, Kuhn JH. "Förslag: Skapa ett megataxonomiskt ramverk som fyller alla huvudtaxonomiska led, för riket Riboviria" . Internationella kommittén för taxonomi för virus (ICTV) . Hämtad 2020-05-21 .CS1 -underhåll: flera namn: författarlista ( länk )
- ^ Kuhn, Jens H. (2020). "Virustaxonomi". Referensmodul i biovetenskap . Encyclopedia of Virology . s. 28–37. doi : 10.1016/B978-0-12-809633-8.21231-4 . ISBN 978-0-12-809633-8. PMC 7157452 .
- ^ STRAUSS, JAMES H .; STRAUSS, ELLEN G. (2008). "Subvirala agenter". Virus och mänskliga sjukdomar . Elsevier. s. 345–368. doi : 10.1016/b978-0-12-373741-0.50012-x . ISBN 978-0-12-373741-0. S2CID 80872659 .
- ^ TaxoProp 2015.002aG
- ^ "80.002 Avsunviroidae - ICTVdB Index of Virus." (Webbplats.) US National Institutes of Health webbplats. Hämtad 2007-09-27.
- ^ "80.001 Popsiviroidae - ICTVdB -virusindex." (Webbplats.) US National Institutes of Health webbplats. Hämtad 2007-09-27.
- ^ Krupovic, Mart; Kuhn, Jens H .; Fischer, Matthias G. (7 oktober 2015). "Ett klassificeringssystem för virofager och satellitvirus" . Arkiv för virologi . 161 (1): 233–247. doi : 10.1007/s00705-015-2622-9 . PMID 26446887 .